Heat map (données OMICS) dans Excel, tutoriel
Ce tutoriel explique comment calculer et interpréter Heat map (données OMICS) avec Excel en utilisant XLSTAT.
Jeu de données pour construire une heat map avec XLSTAT
Pour ce tutoriel, nous utilisons un tableau de données correspondant à 1847 protéines quantifiées sur 19 échantillons d’une feuille de maïs en fonction de 4 méthodes d’extraction basées sur un label-free shotgun proteomics (Langella et al. 2013). Nous remercions chaleureusement la plateforme PAPPSO (Gif-Sur-Yvette, France) d’avoir mis à disposition ce jeu de données et autorisé de l’utiliser pour ce tutoriel.
Les protéines sont rangées en lignes et les échantillons en colonnes.
But de ce tutoriel
Le but de ce tutoriel est d’utiliser la heat map, un outil exploratoire d’analyse de données, afin d’examiner simultanément une classification de caractères (protéines dans notre cas) et d’échantillons. Nous pourrons également détecter d’éventuels regroupements d’individus similaires caractérisés par des regroupements de caractères corrélés.
Paramétrage d’une heat map avec XLSTAT
-
Pour lancer une analyse heat map avec XLSTAT, cliquer sur OMICs / Heat maps.
-
Dans l’onglet Général, sélectionner le tableau de données dans le champ Tableau caractères/individus. Ici, les individus sont représentés par nos échantillons. L’option caractères en lignes ne doit pas être modifiée, car les protéines sont rangées en lignes au sein du tableau.

-
Dans l’onglet Options, activer l’option filtrage non-spécifique, sélectionner Écart interquartile< et introduisez un seuil de 0.25. Cette option éliminera toutes les protéines dont l’écart interquartile est inférieur à 0.25 (variabilité faible). Ceci améliorera la lisibilité de la heat map.
-
Dans l’onglet Données manquantes, paramétrer une estimation des données manquantes par la méthode du plus proche voisin.
-
Dans l’onglet Graphiques, choisir l’échelle de couleur de la heat map et modifier les options largeur et hauteur afin d’optimiser la taille du graphique.
Analyse des sorties d’une Heat map avec XLSTAT
Tout d’abord, notons que le filtrage non-spécifique a éliminé 1597 protéines en amont de la construction de la heat map.

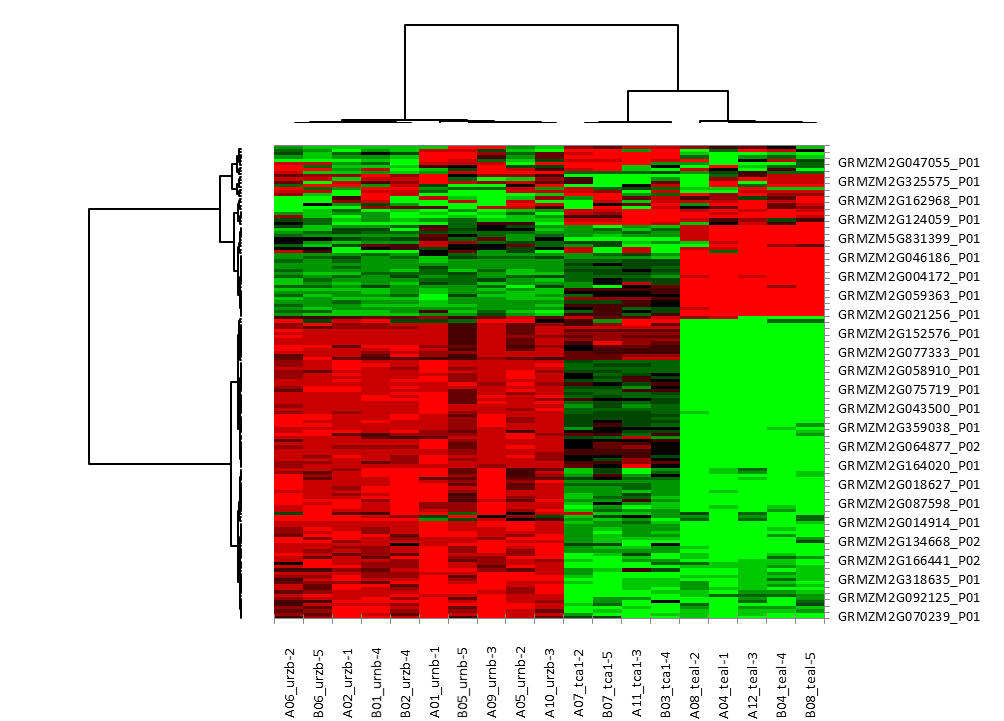
Heat map : les protéines sont classées en lignes et les échantillons en colonnes.
Si nous analysons chaque dendrogramme à part, nous voyons clairement que :
-
Les protéines sont divisées en deux groupes (dendrogramme de gauche) correspondant grossièrement aux protéines membranaires d’une part, et les autres protéines d’autre part.
-
Les échantillons sont divisés en trois groupes (dendrogramme du dessus). Le grand groupe à gauche correspond à des échantillons extraits avec les méthodes URZB et URNB. Le groupe du milieu comprend les échantillons quantifiés avec la méthode TCA1, et le dernier groupe inclut les échantillons extraits avec la méthode TEAL.
Les couleurs de la heat map représentent les valeurs du tableau de données permutées en fonction de l’ordre généré par les dendrogrammes.
Focalisons-nous à présent sur les rectangles / carrés formés à l’intérieur de la heat map.
-
Les grands rectangles vert et rouge à gauche montrent que les échantillons extraits avec les méthodes URZB et URNB sont associés à une expression élevée du groupe de protéines du dessus, en comparaison au groupe de protéines du dessous.
-
Les échantillons TEAL (droite de la heat map) affichent un motif inverse (faible production relative du groupe de protéines du dessus et production relative importante pour les protéines du dessous).
-
Enfin, les échantillons TCA1 (groupement du milieu sur le dendrogramme) semblent associés à des quantités intermédiaires pour la plupart des protéines. Cependant, les protéines en bas de la heat map sont relativement plus abondantes que les protéines du haut).
Bibliographie
Langella O, Valot B, Jacob D, Balliau T, Flores R, Hoogland C, Joets J, Zivy M(2013) Management and dissemination of MS proteomic data with PROTICdb: example of a quantitative comparison between methods of protein extraction, Proteomics. 2013 May;13(9):1457-66.
Cet article vous a t-il été utile ?
- Oui
- Non